Anreicherung von nicht-methylierter DNA leicht gemacht!
Die Analyse des Mikrobioms (z.B. durch Next-Gen-Seq oder PCR) ist immer noch eine echte Herausforderung, da die mikrobielle DNA extrem stark durch die DNA des eukaryotischen Wirts verunreinigt ist. Das neue innovative NEBNext® Microbiome DNA Enrichment Kit bietet hier die Lösung! Es entfernt in weniger als 1 Stunde effektiv und zuverlässig bis zu 98% eukaryotischer DNA und reichert so die mikrobielle, nicht-methylierte Proben-DNA stark an! Die Diversität des Mikrobioms bleibt dabei unverändert intakt!
- Effektive Anreicherung von
mikrobieller genomischer DNA, die mit eukaryotischer DNA
verunreinigt ist (z.B. Speichelproben, Patientenabstriche, Lebens-/Futtermittelproben etc.)
- Einfaches und schnelles Protokoll in weniger als 1 Stunde
- Extrem zuverlässige Ergebnisse
- 16S RNA qPCR Kontroll-Primer im Kit enthalten
Methode:
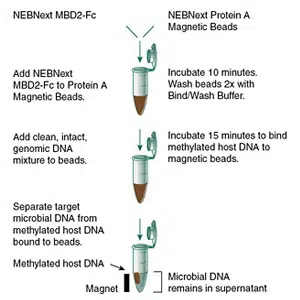
- Präparation der genomischen DNA: Die DNA sollte frei von Proteinen, Proteinase A, SDS und anderen organischen Lösungsmitteln sein; für optimale Ausbeuten sollte die Größe der DNA über 15 kb liegen.
- MBD2-Fc und Magnetische Beads werden in 1x Bind/Wash Buffer zusammen gegeben. Die Reaktion wird 10 Minuten bei Raumtemperatur inkubiert; anschließend werden die Beads zweimal mit Bind/Wash Buffer gewaschen.
- Zugabe der DNA zu den MBD2-Fc Beads. Die Reaktion wird schüttelnd für 15 Minuten bei Raumtemperatur inkubiert.
- Der Überstand enthält die angereicherte, mikrobielle DNA; die Wirts-DNA ist an die Beads gebunden.
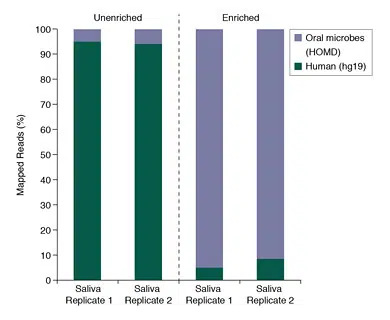
Aufreinigung mikrobieller DNA aus Speichelproben
Die DNA wurde aus DNA von Humanen Speichelproben (Innovative Research) aufgereinigt, und der mikrobielle Anteil wird mit Hilfe des NEBNext Microbiome DNA Enrichment Kit angereichert. Anschließend wurden Sequenzier-Libraries aus angereicherten und nicht-angereichterten DNA Proben hergestellt und auf der SOLiD 4 Plattform sequenziert.
Die Abbildung zeigt den Anteil von 500M-537M SOLiD4 50bp Reads, die entweder auf die Humane Referenz-Sequenz (hg19) oder auf einen mikrobiellen Organismus in der Human Oral Microbiome Database (HOMD) gemappt wurden (da die HOMD Sammlung nicht alle vorkommenden Mikroorganismen umfasst können ca. 80% der angereicherten Proben keiner Referenz zugeordnet werden).
Die Reads wurden mittels Bowtie 0.12.7 und Standard-Einstellungen gemappt.
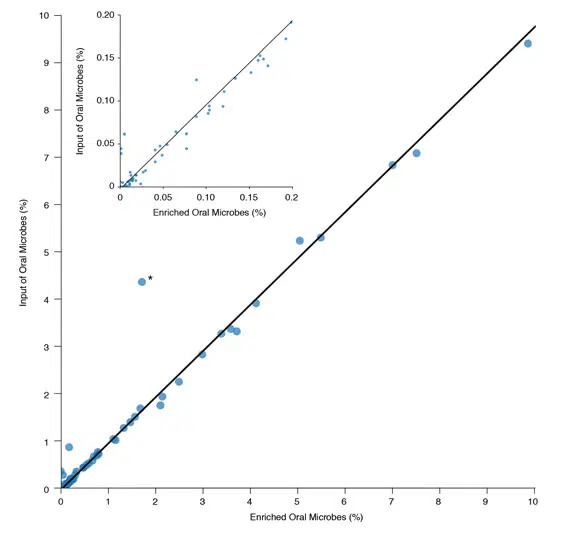
Mikrobielle Diversität
Die mikrobielle Diversität bleibt nach Anreicherung mit dem NEBNext Microbiome DNA Enrichment Kit erhalten.
Die DNA wurde aus DNA von Humanen Speichelproben (Innovative Research) aufgereinigt, und der mikrobielle Anteil wird mit Hilfe des NEBNext Microbiome DNA Enrichment Kit angereichert. Anschließend wurden Sequenzier-Libraries aus angereicherten und nicht-angereichterten DNA Proben hergestellt und auf der SOLiD 4 Plattform sequenziert.
Die Abbildung zeigt einen Vergleich der relativen Häufigkeit jeder bakteriellen Spezies aus der HOMD vor und nach Anreicherung mit dem NEBNext Microbiome DNA Enrichment Kit.
Die Häufigkeit wurde aus der Anzahl der Reads die pro Spezies gemappt werden konnten abgeleitet und ins Verhältnis zu allen Reads, die auf die HOMD gemappt werden konnten, gesetzt.
Es wurden 501M 50bp SOliD4 Reads der angereicherten Proben mit 537M 50bp SOLiD4 Reads der unangereicherten Proben verglichen. Die Reads wurden mittels Bowtie 0.12.7 und Standard-Einstellungen gemappt.
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.