EpiMark Methylated DNA Enrichment Kit
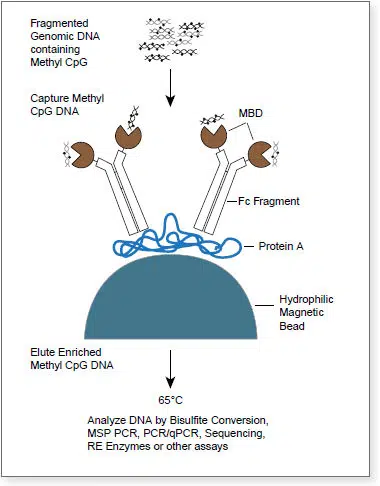
Mit dem EpiMark Methylated DNA Enrichment Kit können Sie zuverlässig und selektiv CpG methylierte dsDNA für subsequente Analysen anreichern. Das Kit nutzt dabei die methyl-CpG bindende Domäne des humanen MBD2a Proteins als Köder. Zur einfachen Aufreinigung ist das MBD2a-Protein mit dem Fc-Teil des humanen IgG1 fusioniert und kann daher leicht mit magnetischen Protein A Beads präzipitiert werden. So erhalten Sie nach dem Pull-down eine spezifisch-angereicherte doppelsträngige, methyl-CpG-haltige DNA. Durch die optimale Abstimmung der Kit Komponenten erreichen Sie eine ausgezeichnete Sensitivität und Selektivität.
- Exzellente Sensitivität auch bei geringer DNA-Input Menge
- Einfaches, schnelles und reproduzierbares Protokoll für angereicherte Methyl-DNA in weniger als 2 Stunden
- Die ideale Methode in Kombination mit weiteren subsequenten Analysen, z.B. Next-Gen-Sequenzierung oder Bisulfit-Konvertierung
Das EpiMark Methylated DNA Enrichment Kit Kit enthält alle notwendigen Komponenten und Puffer für eine schnelle und selektive Anreicherung in weniger als 2 Stunden. Die angereicherte methylierte DNA kann anschließend direkt in diversen Applikationen, z.B. Adaptor-Ligation für Next-Generation-Sequencing eingesetzt werden.
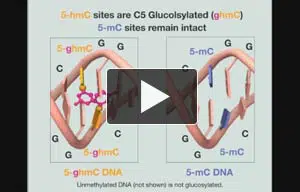
EpiMark 5-mC and 5-hmC Analysis Kit
Das konkurrenzlose, PCR-basierte Analyse Kit zur einfachen Quantifizierung von 5-mC und 5-hmC!
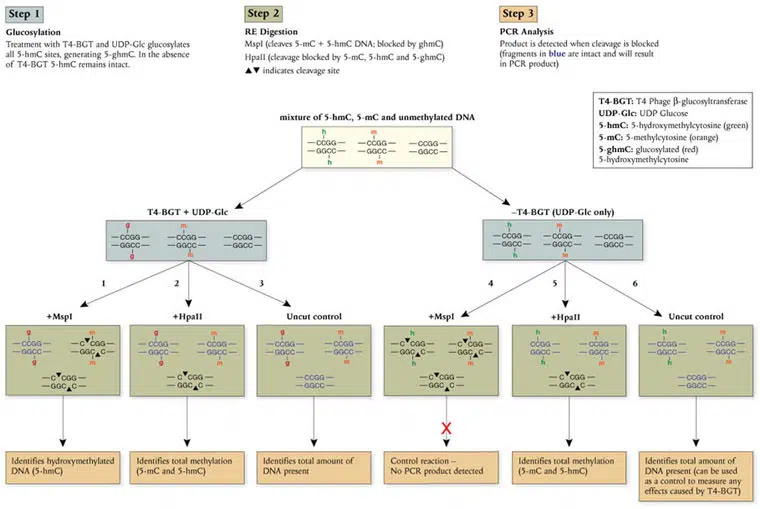
Mit dem EpiMark® 5-hmC and 5-mC Analysis Kit präsentieren wir Ihnen einen echten Durchbruch in der Epigenetik-Forschung. Das innovative und zuverlässige Kit basiert auf dem DNA Verdau mit den methylierungsabhängigen Restriktionsenzymen MspI und HpaII und anschließender PCR-Analyse (Endpunkt oder real time).
- Zuverlässiges und reproduzierbares Kit-Format
- PCR/qPCR-basierte Identifikation bzw. Quantifizierung von 5-mC und 5-hmC
- Einfaches 3-Schritt Protokoll
- Ideal für Hochdurchsatz-Experimente
Mit einem einfachen 3-Schritt-Protokoll identifizieren und quantifizieren Sie so in einem DNA Locus Ihrer Wahl den Methylierungszustand sowohl für 5-Methyl-Cytosin (5-mC) als auch 5-Hydroxymethyl-Cytosin (5-hmC)
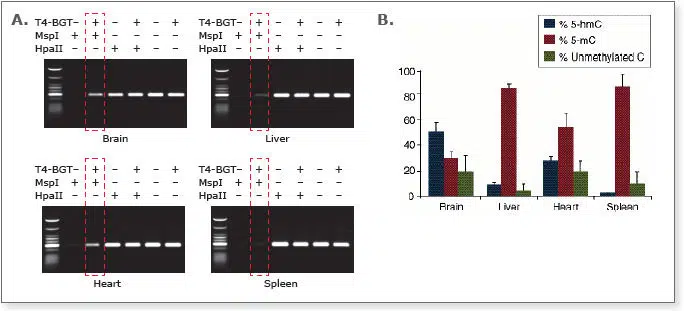
A) Typische EpiMark Analyse zur Untersuchung der DNA Methylierung (PCR-Analyse) eines Gens an gDNA Proben aus 4 verschiedenen Gewebearten eines Individuums (hier Locus 12 einer BALB/C Maus): nur 6 PCR Reaktionen werden für eine eindeutige Methylierungsanalyse benötigt. Das gestrichelte rote Kästchen verdeutlicht das Auftreten von 5-hmC in den jeweiligen Gewebearten.
B) Typische EpiMark Analyse in der real time PCR (qPCR). Mittels qPCR wurde die Menge an 5-hmC und 5-mC in den DNA-Proben aus unterschiedlichen Gewebearten bestimmt. Die exemplarischen Daten belegen eindrucksvoll eine gewebespezifische Verteilung von 5-hmC bzw. 5-mC.
EpiMark Bisulfite Conversion Kit
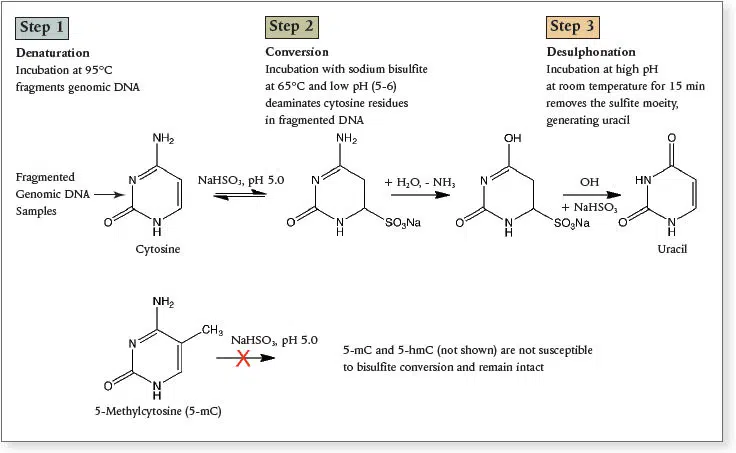
Die Sequenzierung von Bisulfit-behandelter DNA ist zur Zeit die meistverwendete Methode und somit der Goldstandard zur Analyse von DNA-Methylierungen. Hierbei wird die Ziel-DNA fragmentiert, mit Natrium-Bisulfit bei pH 5-6 und 65°C inkubiert und anschließend bei RT für 15 min im basischen Milieu desulphoniert. So werden letztendlich alle nicht-methylierten Cytosine der Ziel-DNA chemisch desaminiert und zu Uracil konvertiert. 5-mC und 5-hmC können mit dieser Behandlung jedoch nicht konvertiert werden und bleiben intakt.
- Garantiert vollständige Konvertierung aller unmodifizierten Cytosine zu Uracil
- enthält alle notwendigen Reagenzien, Puffer, DNA-Aufreinigungssäulchen in optimierten und abgestimmten Formulierungen
- Einfaches, schnelles und reproduzierbares Protokoll – auch für Einsteiger geeignet
Durch eine einfache Sequenzierung der konvertierten DNA und anschließendem Vergleich mit einer unbehandelten Referenz-DNA kann die C zu U Konversion exakt analysiert werden. Das neue EpiMark® Bisulfite Conversion Kit erleichtert die komplexen Arbeitsschritte und umfasst alle notwendigen Reagenzien und Puffer für zuverlässige und reproduzierbare Bisulfit-Analysen.
Das EpiMark® Bisulfite Conversion Kit erleichtert die komplexen Arbeitsschritte der Cytosin/Uracil-Konvertierung methylierter DNA. Dank optimierter Protokolle und der im Kit mitgelieferten Reagenzien, Puffer und DNA-Aufreinigungssäulchen erhalten Sie garantiert und zuverlässig eine 100%ige Konvertierung für reproduzierbare Ergebnisse. So macht das EpiMark® Bisulfite Conversion Kit eine Bisulfit-Konvertierung auch für Einsteiger zum Kinderspiel!
Vollständige DNA-Konvertierung:
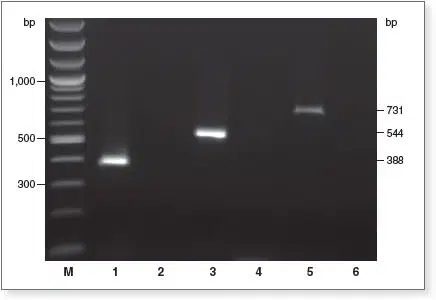
1 µg gDNA wurde mit dem Epimark Bisulfite Conversion Kit behandelt und mit der EpiMark Hot Start Taq analysiert: Spur 1, 3, und 5 sind die erwarteten Amplifikationen mit PCR-Primern spezifisch für Bisulfit-konvertierte DNA; Spur 2, 4, und 6 PCRs sind die Kontrollen mit Primern spezfisch für unbehandelte DNA. Das Ausbleiben der Amplifikation in diesen Spuren belegt eine vollständige Konvertierung der gDNA durch das Epimark Bisulfite Conversion Kit!
EpiMark Hot Start DNA Polymerase
EpiMark® Hot Start Taq DNA Polymerase – optimiert für „Bisulfit-PCRs“
Für für bisulfit-konvertierte DNA gibt es keine bessere PCR Polymerase als die neue EpiMark Hot Start DNA Polymerase von NEB! Ein optimierter, stabilisierender Puffer und ein modifiziertes Enzym machen hier den Unterschied und erlauben eine zuverlässige Amplifikation.
- Spezielle Puffer-Formulierung optimiert für PCR an Bisulfit-konvertierter DNA
- Aptamer-modifizierte Hot Start Taq Polymerase ohne Hitzeaktivierung schont Bisulfit-DNA
- 68°C Extension-Temperatur für optimale Perfomance auf Bisulfit-DNA
EpiMark Nucleosome Assembly Kit
In der Reihe der EpiMark® validierten Reagenzien für Epigenetik-Studien präsentieren wir Ihnen das EpiMark Nucleosome Assembly Kit. Es enthält alle notwendigen Komponenten, um mit Ihrer Ziel-DNA oder der beiliegenden Kontroll-DNA unmodifizierte, rekombinante humane Nukleosomen zu bilden. Die assemblierten Nukleosomen dienen als bevorzugtes Substrat für anschließende ChIP oder HAT- Assays oder für in vitro Studien mit DNA modifizierenden Enzymen (Acetylasen, Methylasen, Chromatin-bindende Proteine etc.).
- Hochreines, rekombinantes System aus formierten Histon-Dimeren / Tetrameren
- Vereinfacht die in vitro Oktamer-Formation
- Einfaches und schnelles Verdünnungsprotokoll für eine Assemblierung in wenigen Stunden
Prinzip des Nucleosome Assembly Kits
Die in vitro Nukleosom Assemblierung zum funktionalen Histon-Oktamer ist mit den neuen Kit besonders schnell, einfach und zuverlässig darstellbar: Sie mischen lediglich in einem Hochsalz-Puffer die vorgefertigten H2A/H2B Dimere und H3.1/H4 Tetramere mit der von Ihnen gewünschten DNA. Während der anschließenden Dialyse gegen einen Niedrigsalzpuffer bildet sich das funktionale Nukleosom aus Histon-Oktamer und DNA – fertig!
So erhalten Sie pro Reaktion in nur wenigen Stunden Ausbeuten von ca. 50 pmol Nukleosomen (mit beliegender 208 bp Kontroll-DNA). Die erfolgreiche Assemblierung können Sie mit einem einfachen Gel-Shift Assay (Protokoll beiliegend) überprüfen.
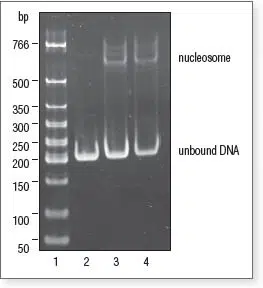
Gel Shift Assay: Proben der Nucleosome Assembly Reaktionen wurden auf einem 6% Polyacrylamid-Gel in 0.5X TBE analysiert.
Spur 1: Low Molecular Weight DNA Ladder (NEB #N3233)
Spur 2: Nucleosome Control DNA.
Spur 3: Ansatz mit Oktamer*-zu-DNA-Verhältnis von 0,5:1
Spur 4: Ansatz mit Oktamer*-zu-DNA-Verhältnis von 1:1
*Oktamer = 2:1 Mischung von Histon H2A/H2B Dimer und Histon H3.1/H4 Tetramer.
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.