Der Industrie-Standard für die High Fidelity PCR
Die Q5 High-Fidelity DNA Polymerase setzt die Maßstäbe für Proofreading Polymerasen und verbindet extreme Genauigkeit mit höchster Zuverlässigkeit!
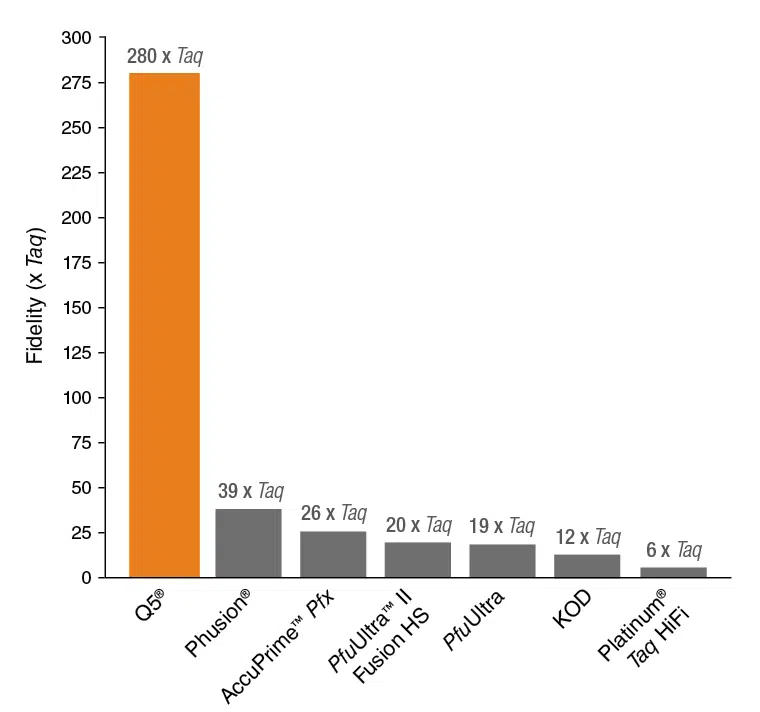
Dabei ist die Q5 High-Fidelity DNA Polymerase ~280 mal genauer als Taq DNA Polymerase und sogar über 7 mal genauer als Phusion Polymerase. Damit besitzt die Q5 HiFi Polymerase die beste Fehlerkorrektur aller erhältlichen Proofreading Polymerasen.
Engineered for Performance
Unsere Q5 High-Fidelity DNA Polymerase basiert auf einer einzigartigen Proofreading Polymerase, die zusätzlich mit einer DNA-bindenden Proteindomäne fusioniert wurde.
Die zusätzliche Sso7d DNA binding domain sorgt für eine dramatische Steigerung der Prozessivität und verbessert die Geschwindigkeit, die Genauigkeit sowie die Zuverlässigkeit der PCR-Reaktion.
Video: Q5 High-Fidelity Polymerase
Bestellen Sie jetzt ein kostenfreies Sample der Q5 High Fidelity DNA Polymerase und überzeugen Sie sich selbst!
Die 5 Qualitätsmerkmale der Q5 Polymerase:
Q1 Genauigkeit (Fidelity)
„Ultra-Fidelity“ dank einer Genauigkeit von ~280x höher als Taq DNA Polymerase und ~7x höher als Phusion Polymerase
Q2 Zuverlässigkeit
Exzellente Spezifität und hohe Ausbeuten ohne aufwendige Reaktionsoptimierung
Q3 Einsatzbereich
Überragende Performance auf allen DNA Templates (egal ob AT-reich oder GC-reich)
Q4 Geschwindigkeit
Extrem kurze Extensionszeiten (nur 10 Sekunden/ kb auf Plasmid-DNA)
Q5 Länge der Amplikons
Zuverlässige Amplifikationen bis 20 kb Länge (Plasmid-DNA) und 10 kb auf komplexen gDNAs
Das sagen unsere Kunden zur Q5 High Fidelity DNA Polymerase
|
„Mutageneseprotokolle über Blunt-End Ligationsstrategien funktionierten schnell, mit hoher Ausbeute und ohne erkennbaren Fehler! Top!!! Ich habe meine Strategie zur Mutagenisierung komplett auf Q5 Hot-Start umgestellt!“ Sebastian Franken, Life-Science-Inkubator GmbH, Bonn |
||||
 |
||||
|
„Seit Monaten konnte keine Amplifikation bei einer Overlap PCR erzielt werden. Doch Dank der Q5 Hot Start DNA Polymerase gelang der Schritt sofort. Wir werden uns jederzeit wieder für dieses Produkt entscheiden! Vielen Dank NEB!“ Sonja Windmann, Virologie, Uniklinik Essen |
||||
 |
||||
|
„Die Testung ergab spezifischere Amplifikation (weniger unspezifische Nebenbanden) und vergleichbare oder höhere Ausbeuten.“ Peter Banasik Genexpress GmbH, Berlin |
||||
 |
||||
|
„Bei Sequenzierung bisher noch keine Fehler gefunden!“ N.N. MPI für Entwicklungsbiologie Tübingen |
||||
 |
||||
|
„Wir haben ein ziemlich langes Amplicon (das murine UTY-Gen, ca. 3700 bp), das uns bei der Klonierung und unserem PCR öfter Schwierigkeiten gemacht hat (…). Bisher haben wir immer dafür die Phusion Hot Start II High Fidelity benutzt. Mit dem Q5 Hot Start High Fidelity haben wir jetzt sogar ein besseres Ergebnis erzielt und sparen dabei noch sehr viel Zeit (fast 2 Stunden), denn mit der Phusion hat die PCR-Reaktion des 3700bp-langes Gen ca. 4:13 Stunden gedauert.“ N.N. Hämatologie/Onkologie, Uniklinik Münster |
||||
 |
||||
|
„Ich verwendete für meinen DNA Klonierungen schon seit vielen Jahren Phusion DNA Polymerase. Ich war die meiste Zeit zufrieden, jedoch musste ich leider oftmals erst spezifische Bedingungen etablieren, um mit dieser Polymerase erfolgreiche PCR Reaktionen zu erzielen, was leider sehr viel Zeit in Anspruch genomment hatte. Unlängst probierte ich zum ersten Mal Q5 Polymerase aus, und ich war überrascht, wie PCR Amplifikationen auf Anhieb geklappt haben, die bei Verwendung von Phusion DNA Polymerase nicht funktionierten. Ich kann daher Q5 Polymerase sehr empfehlen!“ Stefan Weitzer, Medical University of Vienna, Center for Medical Biochemistry |
||||
 |
||||
|
„Amplifikation aus Single Worm Nematodenlysat hat gut funktioniert. Dies ist erfreulich, da nicht selbstverständlich.“ N.N. Zoologie, Uni Köln |
||||
 |
||||
|
„Die Hot-Start-Variante der Q5 funktioniert merklich. PCR-Ansätze unter sonst gleichen Bedingungen lieferten mehr Ausbeute bei der Hot-Start-Variante als bei der normalen Q5 oder Phusion.“ N.N. Buchmann Institut, Uni Frankfurt |
||||
 |
||||
|
„Ich habe die Gratismuster an zwei „Sorgenkindern“ meiner Standard PCRs getestet (...) Mit der Q5 Hot Start Polymerase bekam ich endlich zuverlässig und reproduzierbar meine Banden (...) Herzliche Grüße und nochmal besten Dank für die Proben!“ Doris Glass, Physiologie II, Uniklinik Bonn |
||||
 |
||||
|
„Die Polymerase konnte ohne grosse Umstellung des Protokolls verwendet werden (addaptiert zu DreamTaq). Dies ging besser als mit Pfu!“ N.N. Klinische Chemie & Klinische Pharmakologie, Uniklink Bonn |
||||
 |
||||
|
„Im Vergleich zu anderen Polymerasen ist kein unerwünschter Hintergrund (z.B. unerwünschten Banden) aufgetreten und die erwarteten Banden sind eindeutig amplifiziert worden.“ N.N. Biochemie und Biologie, Universität Potsdam |
||||
 |
||||
Die Q5 HiFi Polymerase im Vergleich mit anderen Proofreading Polymerasen
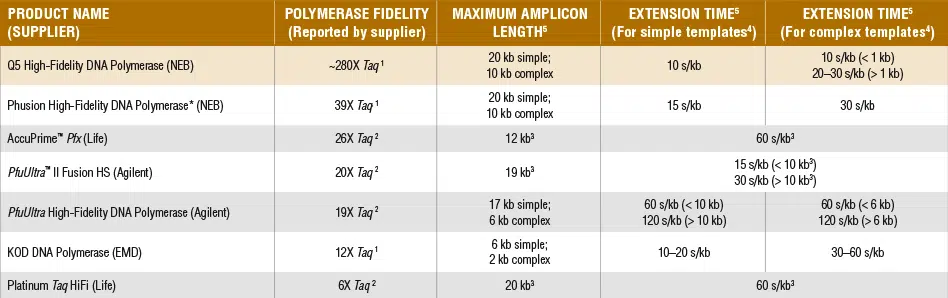
Tabelle: Vergleich von erhältlichen HiFi DNA Polymerasen
1 We continue to investigate improved assays to characterize Q5’s very low error rate to ensure thatwe present the most accurate fidelity data possible (Potapov, V. and Ong, J.L. (2017) PLoS ONE.12(1): e0169774.).
2 PCR-based mutation screening in lacZ (NEB), lacI (Agilent) or rpsL (Life)
3 Template not specified.
4 Simple templates include plasmid, viral and E. coli genomic DNA. Complex templates include plant, human and other mammalian genomic DNA.
5 Values provided by individual manufacturers.
Q5 DNA Polymerasen liefern robuste Ergebnisse unabhängig vom GC Gehalt des Templates
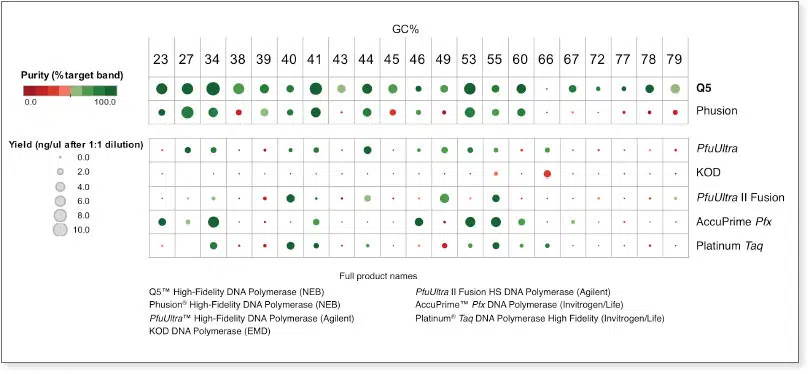
Mit Q5 High-Fidelity DNA Polymerase, Phusion Polymerase und anderen kommerziell erhältlichen Polymerasen wurden unterschiedlichste genomische Targets amplifiziert. Alle Polymerase wurden gemäß den Herstellerangaben verwendet, inkl. der Verwendung spezieller GC-Puffer bzw. -Enhancer. Ausbeute und Reinheit der Produkte wurden quantifiziert und in der Abbildung mittels Farbe und Größe der Punkte dargestellt. Ein großer, dunkelgrüner Punkt stellt die beste Performance dar.
PCR
Empfohlene Produkte:



Erhältliche Q5 Polymerase Produkte
Stand: 01.01.2024
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.



