Golden Gate Assembly (GGA) ist eine Methode zur effektiven, gerichteten Klonierung einzelner oder vieler DNA Fragmente ohne störende zusätzliche Basen an den Fragmentübergängen.
GGA nutzt die Eigenschaft von Typ IIS Restriktionsenzymen, außerhalb ihrer Erkennungssequenz zu schneiden. Beim Schneiden entstehen so definierte Basenüberhänge, die für die gerichtete Ligation Ihrer gewünschten DNA Fragmente in den Vektor genutzt werden. Die ursprüngliche Erkennungssequenz des Typ IIS Restriktionsenzyms ist im ligierten Vektor nicht mehr enthalten, sodass beim GGA Verdau und Ligation zeitgleich in einem Tube erfolgen können!
Nutzen Sie unser praktisches NEBridge Golden Gate Tool online, um die Überhangsequenzen für Ihr Klonierungsvorhaben zu designen. Denn die nötigen Typ IIS Schnittstellen und die komplementären Basen im Ligationsüberhang Ihrer Vektoren und Inserts bringen Sie vorab durch High-Fidelity PCR ein.
- Effizient – schnelle, gerichtete & nahtlose Multiplex-DNA-Klonierung
- Flexibel – > 50 Fragmente unterschiedlicher Größen (< 100 bp bis > 15 kb) & GC-Gehalt
- Innovativ – optimierte, neue Enzymentwicklungen verfügbar für GGA
- Einfach – Primerdesign über NEB Golden Gate Assembly Tool auf unserer Webseite
Golden Gate Assembly ermöglicht so die spezifische Assemblierung mehrerer Fragmente ohne unerwünschte, zusätzliche Basen an den Übergängen. Die Genauigkeit hängt dabei von der Länge der überlappenden Bereiche ab. Daher werden für die Methode solche Typ IIS Restriktionsenzyme bevorzugt, die 4 Basen Überhänge erzeugen, wie BsaI/BsaI-HFv2, BbsI/BbsI-HF, BsmBI-v2 und Esp3I und PaqCI.
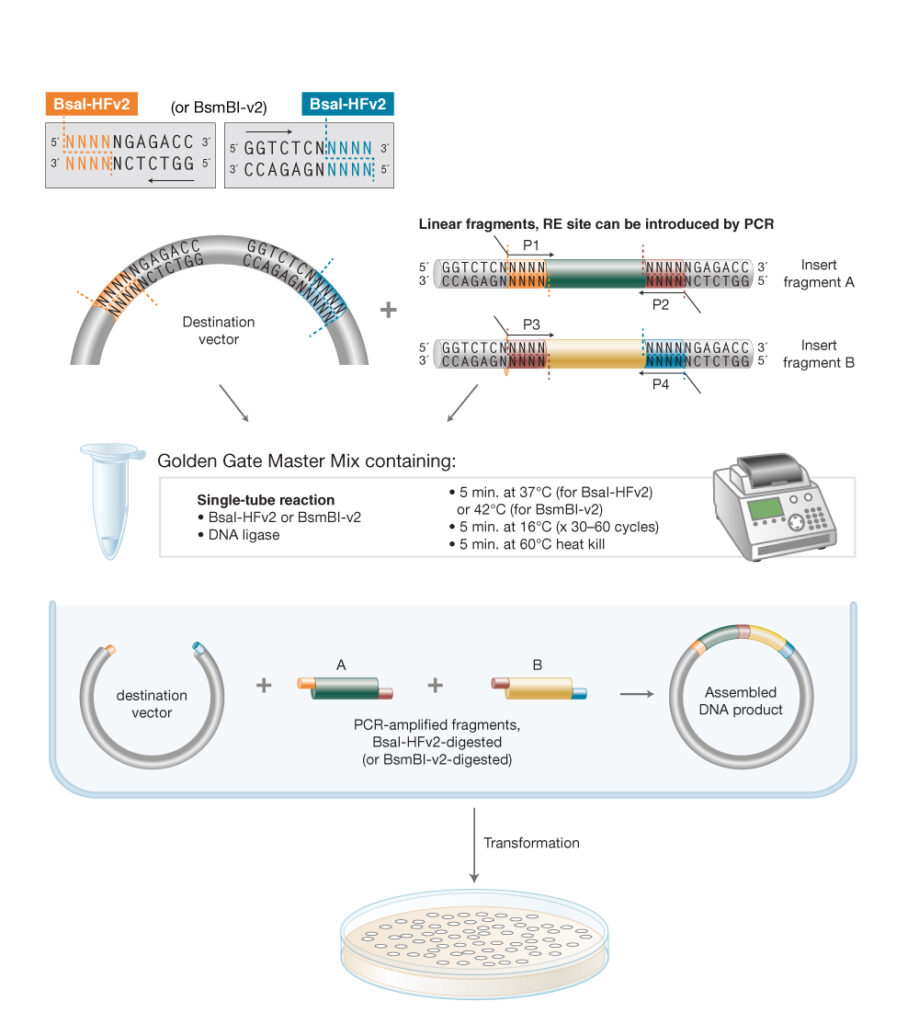
Golden Gate Assembly Workflow
Dank des einfachen Workflows eignet sich Golden Gate Assembly sowohl für einfache als auch komplexe Assemblierungen:
Die Golden Gate Assembly Reaktion läuft als simultane Single-Tube Reaktion ab. Hierzu wird das TypIIS Restriktionsenzym (REase), das außerhalb seiner nicht-palindromischen Erkennungssequenz schneidet, und die T4 DNA Ligase mit den vorbereiteten Inserts und Vektoren zusammengebracht. Die Schnittstellen können vorab mittels PCR Primern integriert werden. Inserts und Vektoren werden so designed, dass die TypIIS Erkennungsstelle distal zur Schnittstelle liegt. Während der Reaktion entfernt die TypIIS REase so die Erkennungssequenz aus dem Assembly und jedes assemblierte Fragment trägt den 3- oder 4-Basen langen komplementären Überhang, der die weitere Assemblierungsreihenfolge steuert. Die Fragmente verbinden sich komplementär, die T4 DNA Ligase schließt die Lücken, und das finale Produkt akkumuliert über die Zeit. Ein Cycling zwischen optimaler Restriktions- und Ligationstemperatur verstärkt die Effektivität der Golden Gate Assemblierung. Golden Gate Assembly kann für die zeitgleiche, gerichtete Assemblierung von 2 bis zu > 50 Fragmenten genutzt werden.
Unser kostenfreies NEBridge Golden Gate Tool hilft Ihnen beim Design der notwendigen Überhänge und stellt sicher, dass die Erkennungssequenz des gewählten Restriktionsenzyms nicht in einer der Zielsequenzen vorkommt.
Über 50 Fragmente gleichzeitig gerichtet klonieren
– Mit NEBs Golden Gate Produkten
NEB ist seit Jahren führend auf dem Gebiet des GGA. Basierend auf vorherigen Forschungsarbeiten zur Optimierung von Type IIS Restriktionsenzymen und DNA Ligasen sowie zum Verständnis der Sequenz-Präferenzen während der Ligation, hat unsere Forschungsgruppe um Greg Lohman kürzlich erstmals ein vollständiges Genom (das des Bakteriophagen T7) aus über 50 synthetisch hergestellten DNA Fragmenten simultan zusammengesetzt!
Damit ist die Tür weit aufgestoßen für neue Einsätze dieser Hochdurchsatz-Technologie.
Das obige Paper basiert auf den zuvor publizierten Arbeiten der gleichen Gruppe, die in der folgenden umfassenden Technical Note prima zusammengefasst sind:
NEB bietet mehr Type IIS Restriktionsenzyme an als andere Anbieter, von denen die meisten auch für Golden Gate Assembly verwendet werden. Informationen zu allen Type IIS Restriktionsenzymen finden Sie in dieser Tabelle. Nutzen Sie gerne die Sortierungsfunktion in den Spaltenüberschriften der Tabelle für einen praktischen Vergleich der Eigenschaften unserer Enzyme.
Unser neustes Mitglied der Typ II S-Restriktionsenzym-Familie – PaqCI
PaqCI, ein AarI Isoschizomer, ist unser neustes Mitglied in der Typ II S-Restriktionsenzym-Familie, das für Golden Gate Assembly verwendet werden kann. Der besondere Vorteil dieses Enzyms ist, dass PaqCI eine Erkennungssequenz von 7 bp hat. Dadurch ist es weniger wahrscheinlich, dass die Erkennungssequenz in der zu assemblierenden DNA-Sequenz bereits vorhanden ist. So wird die Vorbereitung der Schnittstellen an den zu assemblierenden Fragmenten – die sogenannte Domestizierung – zum Kinderspiel.
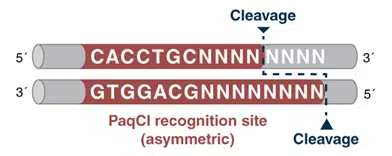
Lesen Sie in unserem Originalartikel mehr über die Besonderheiten von PaqCI und den Einsatzmöglichkeiten im Golden Gate Assembly.
Weitere umfangreiche Informationen zum Golden Gate Assembly finden Sie auch auf unserer USA-Website unter:
Produkte für Golden Gate Assembly
Stand: 01.01.2024
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.