Viele Restriktionsenzyme werden durch DNA Methylierung beeinflusst.
Wenn bestimmte Basen in der Erkennungssequenz modifiziert sind kann die Spaltung der DNA behindert oder vollständig blockiert werden. Die Wissenschaftler von New England Biolabs haben vor einiger Zeit die MspJI Familie der Restriktionsenzyme entdeckt, welche in Abhängig von DNA Methylierung bzw. Hydroxymethylierung DNA schneiden.
Die Enzyme der MspJI Familie schneiden ein 32 bp Fragment um eine zentrale 5-hmC oder 5-mC Base aus. Diese Fragmente können anschließend sequenziert werden, was eine Lokalisierung der Methylierung ohne Bisulfit-Konvertierung möglich macht.
Diese EpiMark validierten Enzyme schaffen neue Möglichkeiten bei der Untersuchung von DNA Methylierungen und insbesondere beim Verständnis der Rolle von 5-Hydroxymethyl-Cytosin.
Auch andere methylierungsabhängige Restriktionsenzyme können für epigenetische Studien eingesetzt werden, wie beispielsweise DpnI und DpnII, welche die gleiche DNA Sequenz abhängig vom Methylierungsstatus erkennen.
- Spezifische Enzyme zur Erkennung von epigenetisch relevanten DNA Modifikationen (5-mC and 5-hmC)
- Einfache Protokolle (Enzymverdau und anschließende Gel-Extraktion)
- Weniger harsch im Vergleich zu Bisulfit-Konvertierung
- Einfache Analyse der Ergebnisse
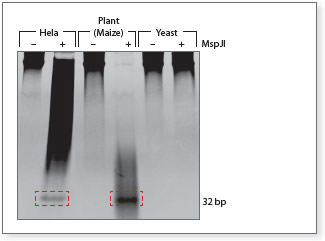
MspJI vereinfacht die Analyse von DNA Methylierungsmustern
MspJI erkennt methylierte und hydroxymethylierte DNA und schneidet ein 32 bp Fragment aus, welches anschließend sequenziert werden kann.
Das Bild zeigt Übernacht-Verdaus von je 1 µg genomischer DNA verschiedenen Ursprungs (DNA aus Hefe weißt keine Methylierung auf).
Produkttabelle
Stand: 01.01.2024
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.