RNA Isolation leicht gemacht
Das „universelle“ Monarch Kit für die einfache RNA Extraktion aus Zellen, Gewebe, Blut, Bakterien, Pflanzen, …
Egal ob Sie RNA aus pflanzlichen Zellen, tierischem Gewebe, menschlichem Blut oder Bakterien isolieren: Sie nutzen dieses Kit für jedes Material und jede Art von RNA – von kurzen miRNAs bis zu rRNAs in voller Länge.
Das Kit vereint die Bindungskapazitäten eines Miniprep Kits mit den Elutionsvolumina eines Kikro-Kits. Auch die Aufreinigung von RNA aus enzymatischen Reaktionen oder einer Trizol-Extraktion sind mit diesem Kit möglich. Die präparierte RNA ist von exzellenter Qualität, mit A260/280- und A260/230-Werten von typischerweise > 2,0, hohen RNA-Integritätswerten und minimalen gDNA-Resten. Somit einigt sich die gereinigte RNA für eine Vielzahl von downstream-Anwendungen wie RT-qPCR, cDNA-Synthese, RNA-seq etc.
- Aufreinigung von bis zu 100 µg hochwertiger Gesamt-RNA aus verschiedenen Probentypen, einschließlich schwer zu lysierender Proben
- Kit enthält Proteinase K für die Probenverarbeitung und Monarch StabiLyse™ DNA/RNA-Puffer für die Probenkonservierung
- Erhalten Sie die Bindungskapazität eines Standard-Mini-Kits (100 µg) mit dem geringen Elutionsvolumen eines Mikro-Kits (10 µl)
- Entfernen Sie genomische DNA über die speziellen Säulen und die enthaltene RNase-freien DNase
- Erhalten Sie den gesamten RNA-Pool Ihrer Probe, einschließlich kleiner RNAs <200 nt
Dabei ist das Monarch Spin RNA Isolation Kit besonders günstig, denn es enthält neben den Säulen und Puffern bereits alle nötigen Reagenzien für die RNA Isolation, wie Proteinase K, DNase I und den Monarch StabiLyse DNA/RNA Buffer zur sicheren und schonenden Lagerung Ihrer Proben.
Bei der Entwicklung aller Monarch Kits wurde auf einen minimalen Umwelteinfluss geachtet
Wir erreichen dies durch den Einsatz besonders dünnwandiger Säulchen und Flaschen. So reduzieren wir den Einsatz von Kunststoff und damit das Müllaufkommen im Labor. Zudem sind alle Komponenten der Verpackung aus recyceltem Material hergestellt.
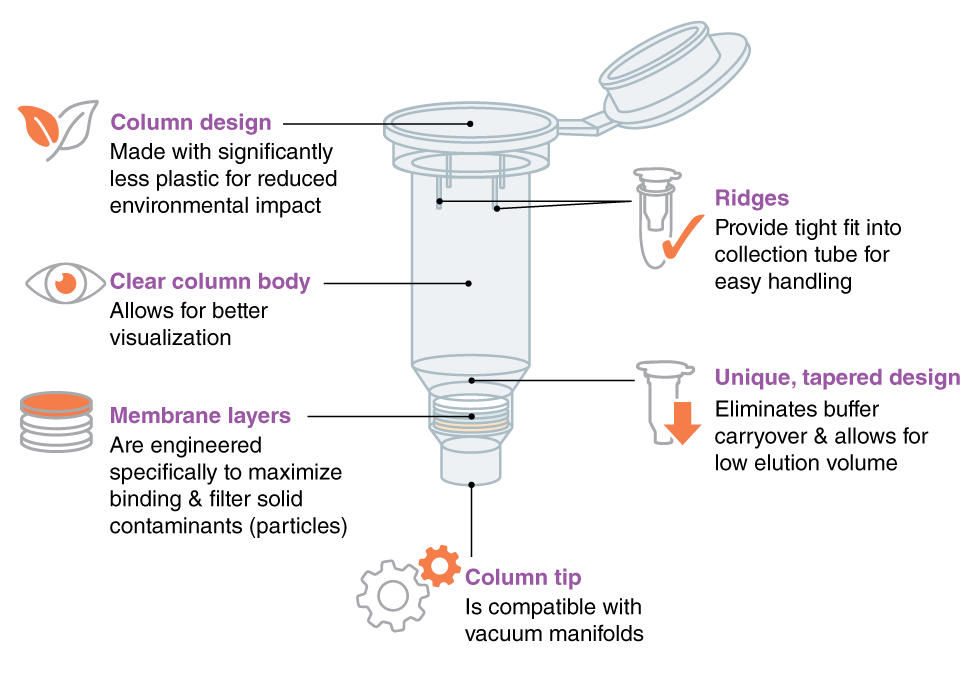
Einzigartiges Säulendesign für saubere Mini Preps
Das einzigartige Säulendesign und der Aufbau der Membran ermöglichen eine qualitativ hochwertige, hochkonzentrierte Nukleinsäureaufreinigung in geringem Elutionsvolumina für eine vielzahl von downstream-Anwendungen. Die Säule wurde mit deutlich weniger Kunststoff hergestellt, um die Umweltbelastung zu reduzieren.
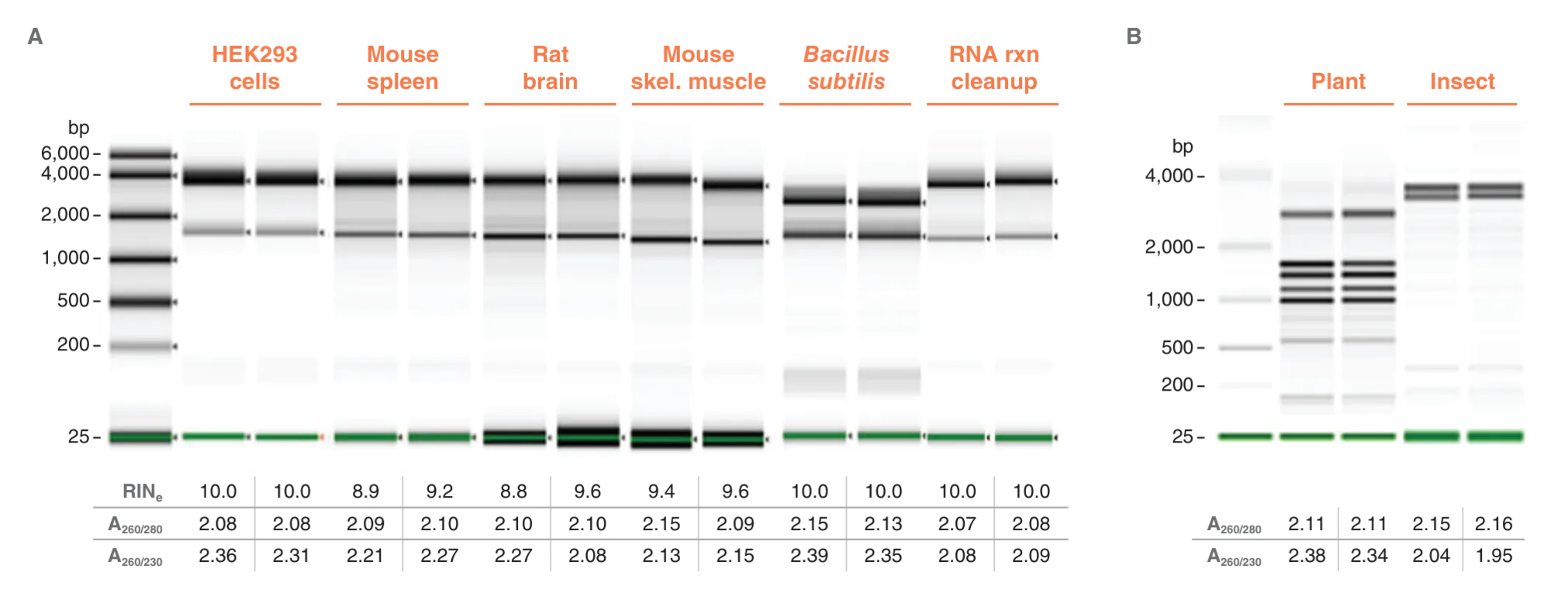
Das Monarch Spin RNA Isolation Kit (Mini) ist eine vielseitige Lösung für die hochwertige RNA-Extraktion aus einer Vielzahl von Probenarten
Die RNA wurde mit dem Monarch Spin RNA Isolation Kit (Mini) aus verschiedenen Ausgangsproben mit unterschiedlichen biologischen Eigenschaften extrahiert, darunter faserreiches, fettreiches und nukleasehaltiges Gewebe von Säugetieren, schwer zu lysierende Proben wie Gram-positive Bakterien, Pflanzen und Insekten sowie Proben aus einer RNA Aufreinigung. Die RNA-Qualität wurde anhand der A260/A280- und A260/A230-Verhältnisse bewertet, die mit einem Mikrovolumenspektrophotometer (Lunatic, Unchained Labs) gemessen wurden. Die RNA-Integrität wurde mit automatisierten Elektrophoresesystemen von Agilent je nach Probentyp gemessen. Für Proben mit bekannten typischen RNA-Profilen wurde das Agilent RNA ScreenTape auf der TapeStation 4200 (A) verwendet. Für Proben mit bekannten atypischen RNA-Profilen, wie z. B. Pflanzengewebe mit Plastiden und Insekten mit ribosomalen Genbruchstücken, wurde der Agilent Bioanalyzer 2100 mit NanoChip verwendet (B).
Das sagen unsere Kunden zu den Monarch RNA Kits:
|
„„Außerdem habe ich auch noch den Monarch Total RNA Miniprep Kit getestet (...) und ich muss sagen ich bin begeistert: funktioniert für unsere Pflanzensamples besser als der RNeasy plant Kit von Qiagen (...). Sogar aus nicht homogenisierten Tomatenwurzeln haben wir eine gute Menge RNA herausbekommen.“ Mitarbeiterin, BOKU, Abteilung für Pflanzenschutz in Tulln |
||||
 |
||||
|
„Currently, a severe but small ssRNA-based plant pathogen, the citrus bark cracking viroid, is threatening hop production in Europe. Since the viroid was discovered in Germany in 2019, we are supporting the viroid monitoring and conducting degradation studies. Therefore, we needed reliable and efficient RNA extractions and compared several extraction protocols including expensive soil extraction kits. Only the Monarch Total RNA Miniprep Kit let to successful RNA extraction in all cases, thus allowing viroid detection from difficult matrices as plant residues, silage, biogas slurry, compost, and even terpenoid-rich fruit skin.“ Dr. Michael Helmut Hagemann, Researcher at the University of Hohenheim, Stuttgart Germany |
||||
 |
||||
Erhältliche Monarch Spin Produkte für die RNA Isolation:
*Produkt nicht als Einzelkomponente verfügbar.
**DNase I (NEB #M0303) kann für den DNase-Verdau auf der Säule und im Reaktionsgefäß verwendet werden. Dabei gelten die gleichen Mengenempfehlungen wie für die rehydrierte Monarch DNase I, lyophilisiert (NEB #T2104).
Stand: 24.03.2025
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.