Eine neue Bisulfit-freie Methode zur Methylom-Analyse
Mangels besserer Alternativen hat sich die Bisulfit Sequenzierung, d.h. Bisulfite Conversion mit anschließender Sequenzierung mittels NGS, zum Goldstandard für die Methylom-Analyse entwickelt. Dabei schädigt diese chemische Behandlung die DNA, was unter anderem eine unerwünschte Fragmentierung der DNA und den Verlust von wertvollem Probenmaterial zur Folge hat. Auch kommt es bei der Bisulfite Conversion zu einer massiven Überrepräsentation AT-reicher so wie zur Unterrepräsentation GC-reicher DNA Abschnitte, wodurch die Auswertung der Sequenzierungsergebnisse erschwert ist.
- Enzymatische Konversion minimiert DNA-Schäden
- Hocheffiziente Library Prep mit konstanten Insertgrößen
- Gleichmäßige GC-Abdeckung & Dinukleotid-Verteilung
- Kombination von E5hmC-seq- & EM-seq-Daten
Fordern Sie jetzt ein Sample kostenfrei an:
Im Gegensatz dazu minimiert die hochwirksame enzymatische Umwandlung der Basen beim NEBNext Enzymatic Methyl-seq Kit die Schädigung der DNA. Im Zusammenspiel mit den mitgelieferten NEBNext Ultra II Library Prep Reagenzien lassen sich so hochwertige NGS Libraries herstellen. Dies ermöglicht einen deutlich besseren Nachweis von 5-mC und 5-hmC bei weniger Sequenzier-Reads.
Video: EM-seq Workflow
Das sagen unsere „Early-Access“ Kunden:
|
„Em-Seq (…) enables us to determine in a precise and DNA sparing way the cytosine methylation status even at low integrity DNA. (…) It also opens new avenues to explorations of methylation at intact long DNA fragments.“ Vladimir Benes, Head Genomics Core Facility at EMBL Heidelberg |
||||
 |
||||
|
„Enzymatic conversion of EM-Seq is THE alternative and our comparisons clearly showed, that the quality of data obtained is better than with conventional bisulfite conversion.“ Dr. Alexander Vogt, Sequencing Specialist, Vienna BioCenter Core Facilities |
||||
 |
||||
NEBNext Enzymatic Methyl-seq Kit:
Erhöhte Sensitivität für den Nachweis von 5mC und 5hmC
Workflow NEBNext Enzymatic Methyl-seq Kit (#E7120) für Illumina-Plattformen:
Die NGS Libraries werden mit den mitgelieferten NEBNext Ultra II Reagenzien und dem optimierten EM-Seq-Adapter erstellt. 10 ng DNA sind dabei als Input ausreichend. Das Enzym TET2 (Tet methylcytosine dioxygenase 2) oxidiert 5-mC und 5-hmC. Dies bietet im nächsten Schritt Schutz vor der Desaminierung durch APOBEC (apolipoprotein B mRNA editing enzyme, catalytic polypeptide-like). Unmodifizierte Cytosine werden von APOBEC zu Uracil desaminiert. Die Libraries werden dann mit einer NEBNext Mastermix-Formulierung der Q5U (einer modifizierten Version der Q5 High-Fidelity DNA Polymerase) amplifiziert und auf einer Illumina Plattform sequenziert.
Das NEBNext Enzymatic Methyl-seq Kit bietet eine hohe und robuste Conversion bei minimierter DNA-Schädigung. In Kombination mit der hocheffizienten Ultra II DNA Library Prep können so mehr CpGs bei weniger Sequencing Reads detektiert werden.
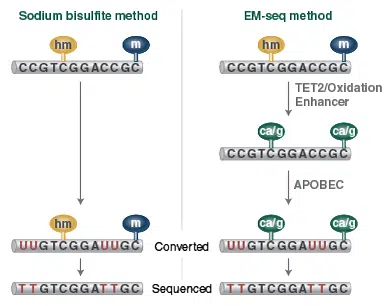
Beim Bilsulfite Sequencing werden die nicht durch Methylierung „geschützten“ Cytosine durch Bisulfite Conversion in Uracil umgewandelt. Bei der NEBNext EM-seq Methode hingegen erfolgt die Umwandlung durch enzymatische Oxidation der methylierten Basen mittels TET2 und einem Oxidation Enhancer. Dadurch sind diese in der anschließenden Desaminierungsreaktion mit APOBEC vor der Umwandlung zu Uracil geschützt. Beide Methoden liefern das gleiche Sequenzierergebnis.
Multiplexing leicht gemacht:
Das NEBNext Enzymatic Methyl-seq Kit (#E7120) enthält bereits Unique Dual Index Primer Pairs für 24-plex Ansätze. Zusammen mit den separat verfügbaren 96-well NEBNext Multiplex Oligos for Enzymatic Methyl-seq (Unique Dual Index Primer Pairs) (#E7140L) können Sie so problemlos bis 120-plex Ansätze realisieren.
Die NEBNext EM-Seq Methode ist für unterschiedliche Sequenzier-Plattformen einsetzbar:
Für Illumina-Plattformen:
Hier wählen Sie das NEBNext Enzymatic Methyl-seq Kit (#E7120), das neben der enzymatischen Methyl-Konvertierung auch die Reagenzien zur schnellen und einfachen Herstellung von Illumina-Libraries enthält.
Andere Sequenzier-Plattformen:
Wir bieten das NEBNext Enzymatic Methyl-seq Conversion Module (#E7125) auch separat an ohne zusätzliche Library Prep Reagenzien an.
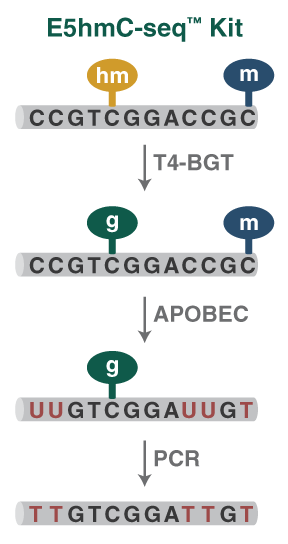
NEBNext Enzymatic 5hmC-seq Kit
Spezifischer Nachweis von 5hmC auf Einzelbasen-Ebene
5-Methylcytosin (5mC) und 5-Hydroxymethylcytosin (5hmC) können durch Sequenzierung von Libraries, die mit dem enzymbasierten NEBNext EM-seq-Workflow oder durch Bisulfit-Konvertierung erzeugt wurden, nachgewiesen werden. Diese Methoden können jedoch nicht zwischen 5mC und 5hmC differenzieren.
NEBNext Enzymatic 5hmC-seq (E5hmC-seq) ist eine enzymbasierte Methode zum spezifischen Nachweis von 5hmC. Diese Methode führt nicht zu DNA-Schäden wie bei der Bisulfit-basierte Methoden und kann ab einer Inputmenge von nur 0,1 ng DNA verwendet werden.
In einem zweistufigen Prozess glycosyliert T4-BGT 5hmC und schützt es so vor einer nachfolgenden Desaminierung durch APOBEC. T4-BGT schützt weder 5mC noch unmethylierte Cytosine, die somit von APOBEC zu Uracil desaminiert werden. Anschließend erfolgt die Amplifikation mit einer NEBNext-Mastermix-Formulierung von Q5U (einer modifizierten Version der Q5 High-Fidelity DNA Polymerase) und die Sequenzierung auf der Illumina-Plattform.
Die für EM-seq und Bisulfit-Sequenzierung verwendeten bioinformatischen Analysetools können auch für E5hmC-seq verwendet werden. E5hmC-seq-Daten können von EM-seq-Daten subtrahiert werden, sodass die genaue Position der einzelnen 5mC und 5hmC bestimmt werden kann.
Beachten Sie, dass Index-Primer nicht enthalten sind und separat erworben werden müssen (NEBNext Primers for Epigenetics, NEB #E3392, NEB #E3404).
Vorteile der NEBNext Enzymatic Methyl-seq im Vergleich zu Bisulfite Sequencing
Intakte DNA führt zu längeren Sequencing Reads
Durch die schonendere Behandlung der DNA im EM-seq-Workflow werden DNA Schäden im Vergleich zur harschen Bisulfit Conversion minimiert. Infolgedessen ist die EM-seq-konvertierte DNA intakter als die bisulfit konvertierte DNA, was zu Libraries mit einem höheren Prozentsatz an längeren Inserts führt, wie in unserer Abbildung dargestellt. Dies ermöglicht längere Sequenzier-Reads, was zu mehr Sicherheit beim Mapping führt.
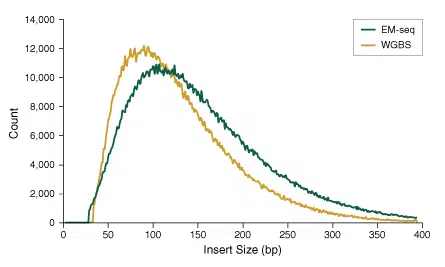
50 ng Humane genomische (NA12878) DNA wurde mit einem Covaris S2 Instrument auf 300 bp geschert und als Input für die EM-seq- und WGBS-Protokolle verwendet. Beim WGBS (Whole Genome Bisulfite Sequencing) wurde das NEBNext Ultra II DNA Library Prep Kit verwendet. Anschließend wurde die Bisulfite Conversion mit dem Zymo Research EZ DNA Methylation-Gold Kit durchgeführt. Die Libraries wurden auf einem Illumina MiSeq® (2 x 76 Basen) sequenziert und die Insert Größen mit Picard 2.18.14 bestimmt. Die normierte Häufigkeit jeder Insert-Größe wurde aufgetragen. Sichtbar ist, dass die Insert-Größen bei der EM-seq Methode größer sind. Die DNA wird bei dieser Methode deutlich weniger beschädigt als bei WGBS.
Höhere Library Ausbeuten
Die durch die Bisulfit Conversion entstehenden DNA-Schäden, die Fragmentierung sowie der Verlust an DNA, reduzieren die Library Ausbeute der nach der Amplifikation. Im Gegensatz dazu ermöglicht die schonendere Behandlung im EM-seq-Workflow hochwertige Sequenzierlibraries, die effizient amplifiziert werden können. Die Folge ist nicht nur eine höhere Ausbeute, auch lässt sich diese mit weniger PCR-Zyklen erreichen. Dabei zeigen EM-seq Libraries konstant niedrige Duplikatwerte unabhängig von der Inputmenge.
Einheitliche Coverage unabhängig vom GC Gehalt
Während für eine erfolgreiche Sequenzierung sind sowohl die Ausbeute der Library Prep als auch die Qualität der resultierenden Library entscheidend. Da die Bisulfit Conversion auf unmethylierte Cytosine wirkt und diese schädigt, wirkt sich diese harsche Behandlung überproportional auf GC-haltige Regionen aus. Diese kontextspezifischen Schäden, Brüche und Verluste führen dazu, dass in den resultierenden Libraries GC-reiche Regionen unter- und AT-reiche Abschnitte überrepräsentiert sind. Im Gegensatz dazu zeigen EM-seq Libraries eine einheitliche GC-Coverage.
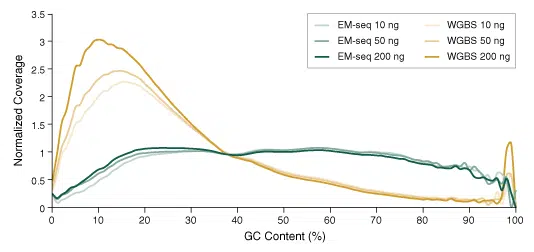
10, 50 und 200 ng Humane Genomische (NA12878) DNA wurden mit einem Covaris S2 Instrument auf 300 bp geschert und als Input in die EM-seq- und WGBS-Protokolle verwendet. Beim WGBS (Whole Genome Bisulfite Sequencing) wurde das NEBNext Ultra II DNA Library Prep Kit verwendet. Anschließend wurde die Bisulfite Conversion mit dem Zymo Research EZ DNA Methylation-Gold Kit durchgeführt. Die Libraries wurden auf einem Illumina NovaSeq 6000 (2 x 100 Basen) sequenziert. Die Messwerte wurden mit bwa-meth 0.2.2.2 auf hg38 aligned. Die GC-Abdeckung wurde mit Picard 2.17.2 analysiert und die Verteilung der normierten Abdeckung über verschiedene GC-Gehalte des Genoms (0-100%) wurde aufgezeichnet. Enzymatic Methyl-seq Libraries haben eine deutlich einheitlichere GC-Coverage und haben nicht die für WGBS Libraries typische AT-Überrepräsentation und GC-Unterrepräsentation.
Alle technischen Details zum NEBNext Enzymatic Methyl seq Kit finden Sie in unserer Technical Note.
NEBNext Enzymatic Methyl-seq Produkte
Stand: 07.06.2024
NEBNext Produkte erhalten Sie zum dauerhaften Niedrigpreis, der nicht weiter rabattiert werden kann.
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.