Hintergrund
Die in vitro RNA-Synthese ist eine wichtige Grundlage der rasant expandierenden RNA Forschung. Die Technik ist vielseitig und erlaubt Ihnen die maßgeschneiderte Synthese des optimalen Transkripts für Ihre Anwendung. Beginnend mit der Herstellung hochqualitativer Templates über die in vitro Transkription bis zur post-transkriptionellen Modifikation:
New England Biolabs versorgt Sie mit einem
vollständigen Set aller notwendigen Reagenzien für die RNA Synthese.

Unsere kostenlose Broschüre
RNA Synthesis –
From Template to Transcript
Feature Article
Minding your caps and tails – considerations for functional mRNA synthesis
Technische Details
Im ersten Schritt der in vitro Synthese stellen Sie ein DNA Template mit der gewünschten Sequenz her. Dabei kann es sich um ein PCR Produkt oder ein Plasmid handeln, das durch Restriktionsverdau linearisiert wurde. Das Template wird dann von einer RNA Polymerase in Anwesenheit von Ribonucleosid-Triphosphaten (rNTP) transkribiert. Das Transkript kann nach der Synthese durch labeling, capping oder 3′ Polyadenylierung weiter prozessiert werden.
Template Herstellung
Die effektive in vitro Transkription beginnt mit einem hochqualitativen Template.
Wenn Sie Plasmid-DNA verwenden, muss diese für ein definiertes Ende des Transkripts vollständig linearisiert sein. Dafür eignen sich die bewährten Restriktionsenzyme von New England Biolabs.
Und wenn Sie von einem PCR-Produkt transkribieren möchten, ist die Q5 High-Fidelity DNA Polymerase die erste Wahl für eine garantiert fehlerfreie Template-Amplifikation.
Kombinierte RNA Synthese und Modifikation
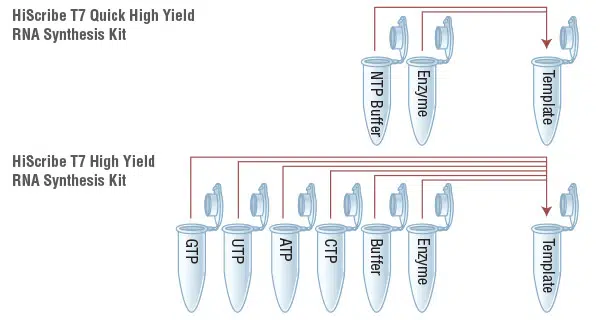
Die beliebten High Yield RNA Synthese Kits von NEB sind optimal für hohe Erträge von bis zu 180 µg/rxn. Sie benötigen lediglich ein T7 (bzw. SP6) Promotor-basierendes dsDNA Template. Den Rest übernehmen unsere Kits. Das HiScribe T7 Quick High Yield RNA Synthesis Kit bietet Ihnen dabei einen effizienten Reaktionsansatz mit nur drei Pipettierschritten (NTP-Puffer Mix, T7 RNA Polymerase und Template-DNA).
Für mehr Flexibilität bieten wir Ihnen das HiScribe T7 High Yield RNA Synthesis Kit an:
Die separat formulierten Nukleotide erlauben dabei die freie Komposition eines benutzerdefinierten Nukleotid-Cocktails, z.B. den Austausch eines Nukleotids gegen ein modifiziertes Analogon.
Co-transkriptionelles Capping:
Biologisch aktive mRNA enthält post-transkriptionelle Modifikationen, wie die 5’Cap Struktur oder die 3’ Polyadenylierung. Diese dienen u.a. zur Stabilisierung und als Bindestelle für Translationsfaktoren.
Jede durch in-vitro-transcription (IVT) hergestellte, neusynthetisierte mRNA muss daher mit den entsprechenden „caps and tails“ versehen werden, soll sie als funktionale Blaupause für eine nachgeschaltete Proteinsynthese dienen.
Dabei haben sich zwei verschiedene mRNA Capping Methoden etabliert: enzymatisch und co-transkriptional.
Viele Forscher nutzen co-transkriptionelles Capping durch den Einsatz von Cap-Struktur Analoga während der in vitro Transkription. NEB bietet dazu diverse Anti Reverse Cap-Analoga (ARCA) und weitere Cap Analoga an. Hilfe bei der Auswahl des für Sie geeigneten Cap Analogs bietet Ihnen unser RNA Cap Analog Selection Chart.
Wir empfehlen aber unsere speziellen HiScribe T7 mRNA Kit with CleanCap Reagent AG oder das HiScribe T7 ARCA mRNA Kit (with tailing), die mit einfachen Workflows, Zuverlässigkeit und hohe Ausbeuten überzeugen.
Praktische Mastermixe im HiScribe T7 Quick High Yield RNA Synthesis Kit (oben). Mehr Flexibilität und Austausch-Möglichkeit von rNTPs im HiScribe T7 High Yield RNA Synthesis Kit (unten).
Schnelle und zuverlässige Protokolle ergeben in gut 2 Stunden hohe mRNA-Erträge mit korrekten Caps und Tails
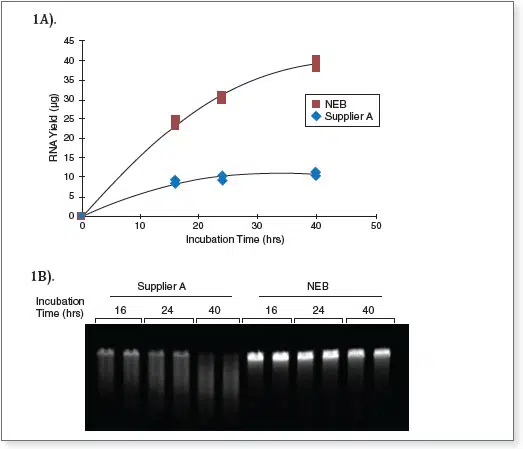
Erhöhte RNA-Ausbeute und Integrität durch zeitlich verlängerte Transkriptions-Reaktionen.
Enzymatisches 5′ Capping
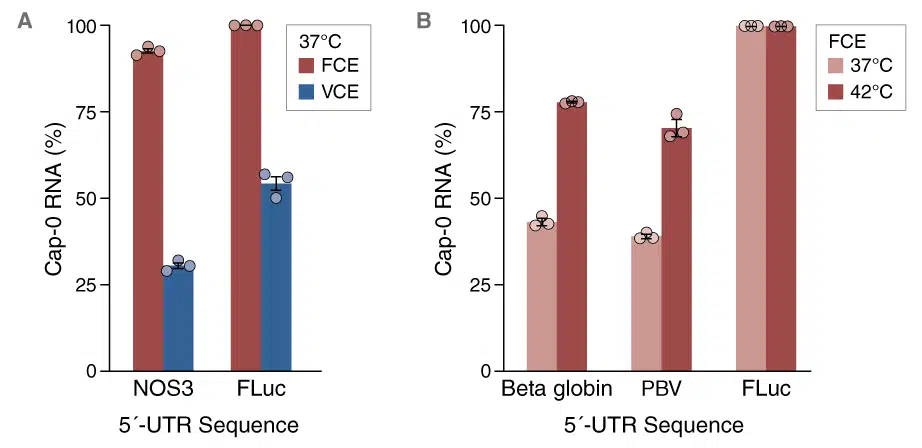
Für das enzymatische Capping empfehlen wir Ihnen das Faustovirus Capping Enzyme (FCE). Durch die verbesserte Effizienz und die hohe Aktivität des Enzyms in einem breiten Temperaturbereich, ermöglicht FCE ein robustes und effizientes Capping auch bei anspruchsvollen Substraten mit Sekundärstrukturen. Auch preislich ist FCE eine attraktive Alternative. Und durch die exzellente Skalierbarkeit birgt es gerade für den Einsatz im Produktionsmaßstab ein erhebliches Potential.
Vergleich der Capping-Effizienz zwischen A) FCE und VCE (Vaccinia Capping Enzyme) und B) FCE mit verschiedenen Substraten und Temperaturen. Weitere detaillierte experimentelle Details finden Sie hier.
Alternativ bieten wir Ihnen das beliebte Vaccinia Capping System. Das 2 Protein-System mit insgesamt drei katalytischen Aktivitäten synthetisiert in unter einer Stunde natürliche Cap0-Strukturen (m7G5’ppp5’N) an nahezu 100% aller in vitro Transkripte. Das Vaccinia-Capping ist kompatibel mit der Cap0- zur Cap1-Umwandlung durch die mRNA Cap 2’-O-Methyltransferase. Die Umwandlung zu Cap1 führt zu einer signifikanten Erhöhung der Translationsausbeute.
RNA Aufreinigung
Nach erfolgreicher in vitro Transkription wird das Template durch DNaseI Verdau abgebaut und das Produkt von DNA-Rückständen befreit. Hier empfehlen wir die DNaseI-XT, die insbesondere in salzhaltigen IVT-Reaktionen der herkömmlichen DNaseI überlegen ist. Wenn Sie mRNA synthetisieren, können Sie zusätzlich Oligo(dT)Beads zur Affinitäts-Reinigung Ihres Produktes verwenden.
mRNA synthesis workflow example & available NEB products
(Klicken Sie auf das jeweilige Kästchen für weitere Produktinfomationen)
Weiterführende Informationen finden Sie unter Technische Ressourcen oder auf neb.com. Informationen zu Namensrechten finden Sie hier.